Reprogramación epigenética no mutacional
La inestabilidad genética es un enabling hallmark, es decir, provoca diversas mutaciones en las células tumorales que promueve a que aumente malignidad del tumor. Años después, se plantea otro enabling hallmark: los cambios de expresión génica en el tumor mediante la modificación de la cromatina, es decir cambios epigenéticos. Estos cambios podrían ser reversibles y temporales en el tumor, y se sospecha que tienen que ver con la resistencia de los tumores.
Esta regulación epigenética se conoce como mecanismo para regular la expresión génica durante el desarrollo embrionario, la diferenciación y la organogénesis. Esta regulación en tumores se puede dar por una indebida metilación de los residuos de citosina en las regiones CpG (normalmente en promotores de genes), por cambios en la modificación postraduccional de las histonas, o por mutaciones en las enzimas que se encargan de modificar estas histonas y metilar o desmetilar el ADN. También se han visto cambios en la expresión génica debido a microARN reguladores (miARN). Todo esto parece tener un papel importante en contribuir a que el tumor gane ciertas características que lo hacen más maligno (aquí puedes saber más sobre cómo afecta en metástasis).
Cuando un tumor aumenta su tamaño, hay zonas del mismo donde no hay vascularización o la que hay es muy pobre, generando una situación de hipoxia. En estas circunstancias, se reduce la actividad de desmetilasas como TET (tet metilcitosina dioxigenasa); estas enzimas catalizan la desmetilación de citosinas para lograr una desmetilación en el ADN. La pérdida de la actividad de estas enzimas genera una hipermetilación en el ADN que, a su vez, causa una pérdida de expresión de algunos genes (como los genes supresores de tumores).
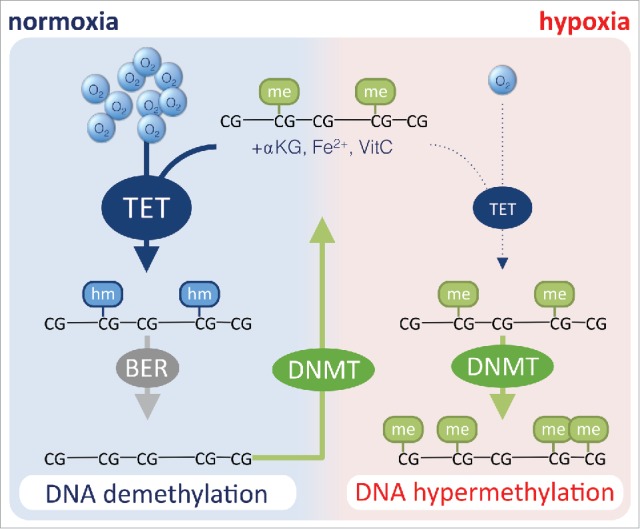
Asimismo, se conoce que en las células tumorales tempranas ocurre una hipometilación que se suele generar por la pérdida o reducción de actividad de la ADN metiltransferasa de mantenimiento DNMT1, lo que causa inestabilidad genómica y aumento de la aneuploidía. Esto se debe a la activación de diversos genes (pueden ser oncogenes) y de transposones que pueden generar mutaciones en más genes.
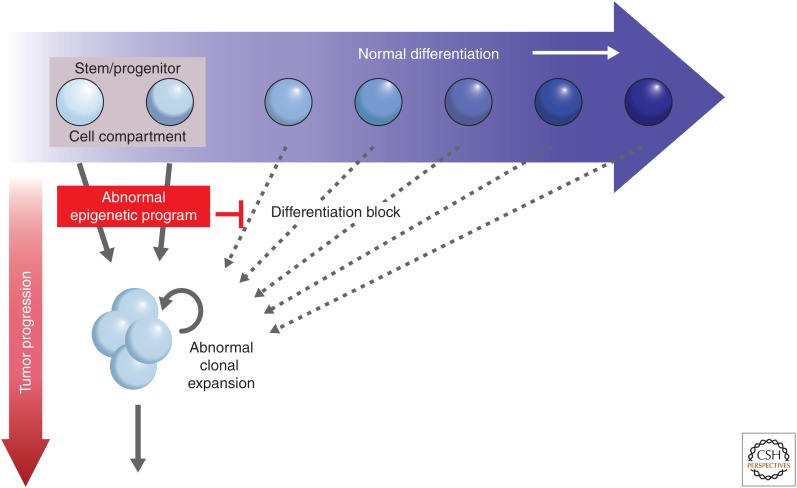
Estas modificaciones epigenéticas, a su vez, suponen un obstáculo para que la célula pueda diferenciarse; de este modo, se mantiene en expansión y puede generar tumores.
Una evidencia sería, por ejemplo, la capacidad reversible de invasión que tienen algunos tumores, que es regulado a su vez por la transición epitelio-mesénquima (EMT). Se ha conocido recientemente un regulador, ZEB1, que induce la expresión de una histona metiltransferasa SETD1B. Esta histona, a su vez, mantiene activo a ZEB1 y, con ello, el estado de invasión. Además, otros factores a los que se exponen las células tumorales (como la hipoxia o citoquinas) pueden inducir EMT y, por tanto, la capacidad invasiva.
También se ha visto que la matriz extracelular cambia entre una célula normal y una tumoral, aumentando la reticulación y la densidad. Así, se generan modificaciones enzimáticas y la composición molecular se altera. Estos cambios provienen de modificaciones en la expresión de genes y no en mutaciones de los mismos.
Por otro lado, una de las características de los tumores es que no todas las células son clones, sino que hay una heterogeneidad genética entre ellas. Se diferencian en distintas mutaciones, pero también se observa la posibilidad de que haya diferencias epigenéticas entre ellas.
Además, se piensa que las células del microambiente tumoral no cambian su función por mutaciones, sino por cambios epigenéticos debido a las señales y factores que recibe del tumor.
Cada vez existen más tecnologías que permiten observar la metilación del ADN en todo el genoma de las distintas células tumorales, además de la modificación de histonas, la accesibilidad a la cromatina e incluso la modificación postranscripcional y traducción del ARN.
La hipermetilación del ADN localizada en las islas CpG es el biomarcador más estudiado. Se han desarrollado varios ensayos bastante sensibles y se ha usado en combinación con el ADN-bisulfito para detectar los niveles de metilación del ADN en pacientes. También se están generando PCR específicas para esta situación donde los primers se unirían a zonas ya metiladas. Una ventaja de estos biomarcadores es que se podría distinguir entre distintos tipos tumorales.
Además, con estos conocimientos surge una nueva terapia conocida como "terapia epigenética" basada en modificar los patrones de metilación tanto de la cromatina como del propio ADN. Algunas de estas terapias serían:
Inhibidores de DNMT: interactúan con las DNMT y forman intermediarios covalentes impidiendo la función de las mismas, es decir, no se metila el ADN.
Inhibidores HDAC: estas enzimas se encargan de eliminar marcas de acetilación en la cola de las histonas.
Se estudia la terapia combinada usando varios medicamentos que afecten a la regulación de la cromatina, pero no se conoce mucho todavía.
Para saber más:
- Epigenetic alterations in cancer. Frontiers in bioscience (Landmark edition) 25, 6, 1058-1109 (2020).
- Epidrugs: targeting epigenetic marks in cancer treatment. Epigenetics 14, 12 (2019).
- Epigenetic Aging: More Than Just a Clock When It Comes to Cancer. Cancer research 80, 3 (2020).
Bibliografía empleada para elaborar el texto y las imágenes:
Baylin SB, Jones PA. Epigenetic Determinants of Cancer. Cold Spring Harbor perspectives in biology 8, 9 a019505. 1 (2016).
Hanahan D. Hallmarks of cancer: new dimensions. Cancer Discov 12, 31-46 (2022).
Thienpont B et al. Tumors smother their epigenome. Molecular & cellular oncology 3, 6 e1240549 (2016).