Insensibilidad a las señales antiproliferativas
En un tejido normal, las señales antiproliferativas mantienen la homeostasis, siendo tanto inhibidores del crecimiento solubles como inhibidores inmovilizados en la matriz extracelular o en la superficie de células cercanas. Estas señales también son percibidas por receptores transmembranales de las células acopladas a circuitos de señalización intracelular. Las señales antiproliferativas pueden bloquear el crecimiento por dos vías distintas:
- Las células pueden ser forzadas a pasar a un estado quiescente (G0) del
que pueden salir en algún futuro por acción de otras señales extracelulares.
- Se puede hacer que las células renuncien permanentemente a su potencial
proliferativo induciéndolas a entrar en estados postmitóticos, normalmente
asociados a la adquisición de rasgos específicos relacionados con la
diferenciación.
Las células cancerosas incipientes deben evadir estas señales antiproliferativas para poder prosperar. Las células controlan y vigilan su entorno externo durante la fase G1 del ciclo celular, y en base a las señales detectadas deciden si proliferan, permanecen inactivas o entran en un estado postmitótico. Las principales señales antiproliferativas son las dadas por los genes supresores de tumores; docenas de ellos actúan en numerosas vías para limitar el crecimiento celular y la proliferación. Los dos prototípicos son RB (pRb) y TP53 (p53), que actúan como nodos centrales para este control; ambas proteínas participarían en una gran red en la que habría redundancia funcional. El producto del gen MF2, Merlin, orquesta la inhibición por contacto acoplando moléculas de adhesión en la superficie celular como la E-cadherina con receptores tirosín quinasa como EGFR, aumentando la adhesión entre células e impidiendo el crecimiento.
Los genes supresores de tumores podrían dividirse en distintas clases según la función primaria de las proteínas que codifiquen:
- Anti-oncogenes: como RB1 y CDKN2A, que codifican respectivamente a pRb y p16INK4A, y antagonizan las actividades promotoras del crecimiento de los oncogenes.
- Genes del checkpoint del daño del ADN: como ATM y TP53, que codifican a ATM y p53, respectivamente, e inducen la muerte celular y senescencia en respuesta al daño celular o al estrés durante la replicación del ADN.
- Genes caretaker: como MUTYH o XPA, que codifican proteínas que ayudan al mantenimiento de la estabilidad genómica.
El gen TP53, que codifica la proteína p53 y está ubicado en el
brazo corto del cromosoma 17 (17p13.1), es el que está más frecuentemente
alterado en los tumores humanos. La proteína p53 es un factor de transcripción
que activa la expresión de múltiples genes diana con papeles esenciales en la
regulación del ciclo celular, la apoptosis y la estabilidad genómica,
recibiendo por ello el nombre de "guardián del genoma". Las principales
mutaciones que sufre en los tumores son de pérdida de función, dándole una
ventaja fundamental a las células cancerígenas: priva a las células de
respuestas supresoras de tumores como la senescencia o la apoptosis, lo que es
una ventaja para su desarrollo (para entender la importancia de la senescencia celular y la apoptosis en el cáncer, pincha en los términos en azul).
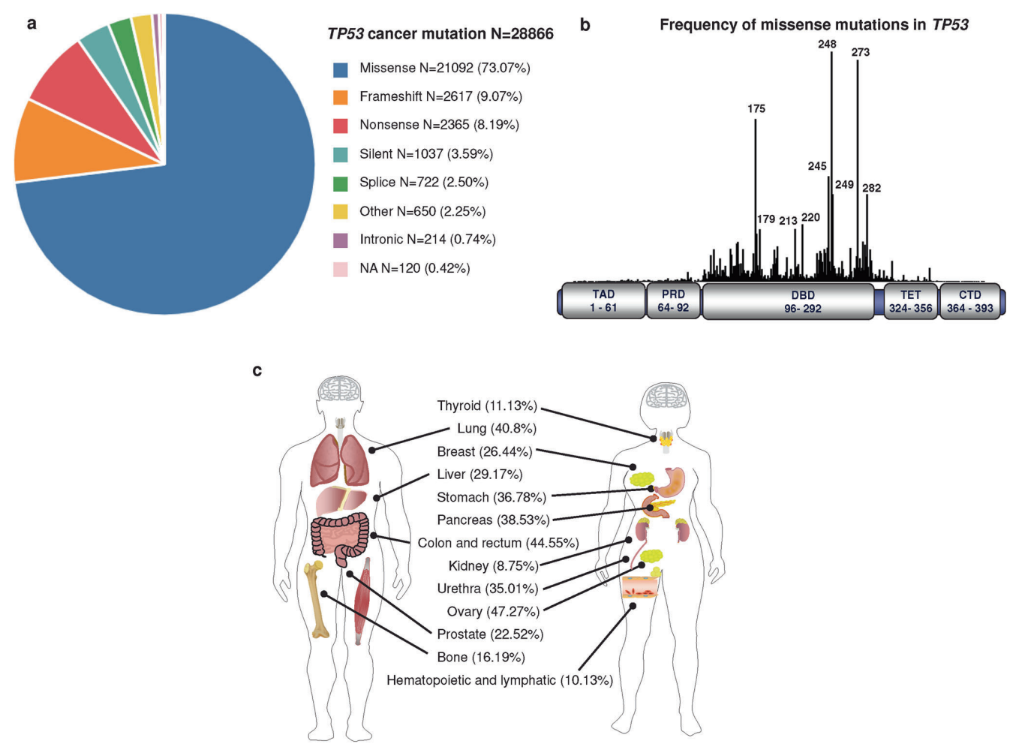
Se ha visto cómo las células tumorigénicas son "adictas" a estas
mutaciones de pérdida de función como las de tipo missense (mutaciones
de cambio de sentido) en el gen TP53; parece que las proteínas p53
mutadas de esta manera permiten que las células tumorigénicas sean capaces de
hacer frente a condiciones limitantes originadas durante la carcinogénesis.
También se ha visto cómo pérdidas del gen TP53 hacen que sea más
probable el desarrollo de tumores.
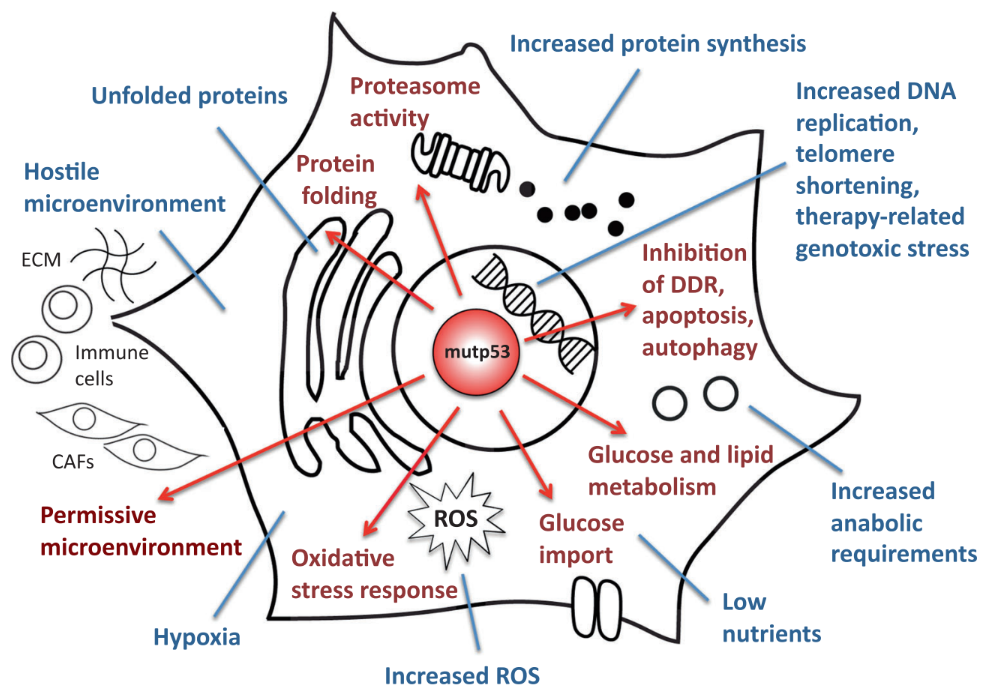
Así, las oncoproteínas p53 mutantes se estabilizan y activan en
respuesta a estímulos de estrés relacionados con el propio tumor y orquestan
mecanismos de respuesta al estrés que facilitan la supervivencia de estas
células tumorales y su adaptación a múltiples condiciones de estrés intrínsecas
y extrínsecas (como estrés genotóxico, oxidativo y proteotóxico, microambiente
tumoral hostil, …). Los procesos de adaptación al estrés inducidos por la
mutación de p53 desencadenan respuestas de feedback positivo que promueven
su propia acumulación en las células cancerosas, fomentando la carcinogénesis.
Las chaperonas de la maquinaria de las Heat-shock protein (Hsp, conformada
por Hsp90, Hsp70 y Hsp40/DNAJA1) promueven la estabilización de p53 mutante. El
sistema Hsp90 está frecuentemente activado de manera aberrante por la
activación de HSF1 en respuesta a múltiples condiciones de estrés (como el
estrés oxidativo, asociado con el crecimiento tumoral y en el que puedes profundizar aquí), por lo que en dichas
circunstancias la proteína p53 mutante va a quedar estabilizada.
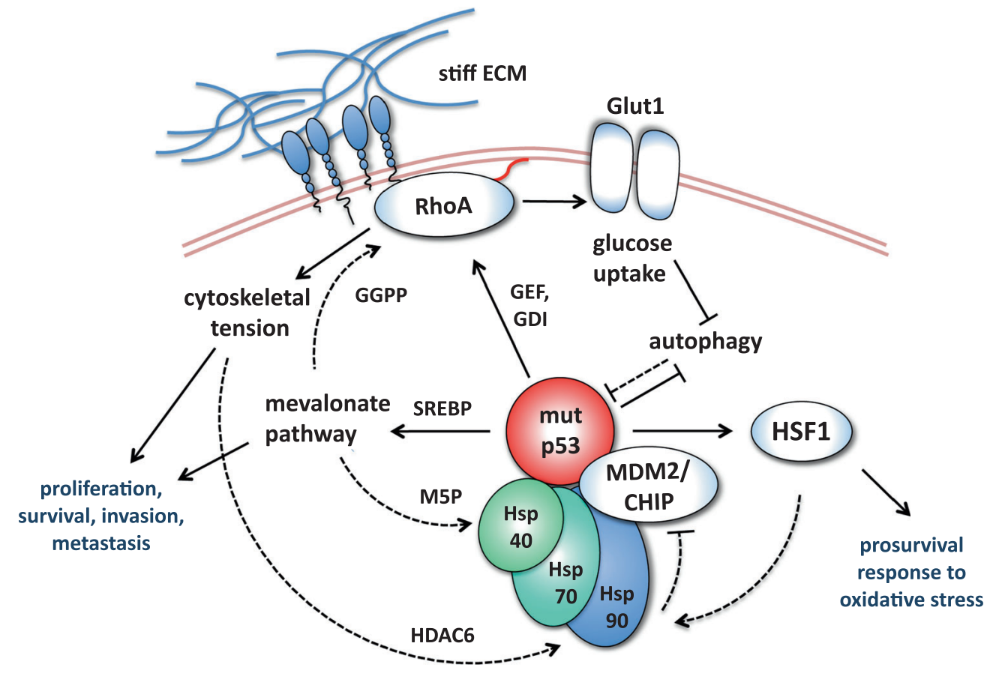
Por tanto, interferir con la acción de esta proteína podría disminuir la proliferación o la supervivencia de los tumores que tengan p53 mutado (la alta frecuencia de mutaciones en TP53 en los tumores lo hace un candidato perfecto para ser usado como diana terapéutica). Hasta el momento, para interferir con la expresión de p53 mutante sólo ha llegado con éxito a las fases clínicas I/II el compuesto APR-246, que revierte la proteína mutante a una conformación similar a la de la proteína sin mutar. Aunque se han hecho multitud de análisis estructurales y bioquímicos en p53, aún se desconocen aspectos de su regulación específica y de su función relacionada con su estructura. MDM2/MDMX es el principal regulador negativo de p53, habiéndose visto que su sobreexpresión está presente en muchos tumores humanos y lleva a la inactivación de p53. Por ello también se están buscando inhibidores que bloqueen la interacción de MDM2 o de MDM2/MDMX con p53 en estas situaciones, además de otro tipo de aproximaciones (como las que evitan la formación de agregados de p53, las que usan anticuerpos frente a p53 o las que son específicas para algunas mutaciones, entre otras), pero todas ellas están aún ensayándose.
En cuanto al gen RB1 (correpresor transcripcional RB 1) fue el primer gen supresor tumoral en ser descubierto y codifica la proteína del retinoblastoma (pRb). Las mutaciones que producen pérdida de función en este gen son la causa principal del retinoblastoma, ya sea familiar o esporádico (por mutaciones somáticas). Aun así, su pérdida de función también tiene relación con una fracción importante de los cánceres más comunes. Además, se vio en los años 90 que había dos genes más que compartían homología de secuencia con RB1: RBL1 (proteína similar a RB 1) y RBL2 (proteína similar a RB 2), que codifican a p107 y p130, respectivamente. Pese a que son similares estructuralmente a pRb y tienen actividad antitumoral, no tienen el papel prominente que posee la proteína del retinoblastoma; de hecho, se ha visto en muchos casos que la actividad antitumoral de p107 y p130 aparece cuando la actividad de pRb está ausente. La supresión de la vía pRb impide que E2F regule el ciclo celular y, por tanto, permite la proliferación celular, lo que hace que las células sean insensibles a los factores que suprimen el crecimiento y que normalmente bloquean el avance a través de la fase G1 del ciclo. También el efecto de TGFβ es importante. Se sabe que previene la fosforilación que inactiva pRb; TGFβ provoca la síntesis de las proteínas p15 y p21, encargadas de bloquear los complejos ciclina:CDK responsables de esta fosforilación.
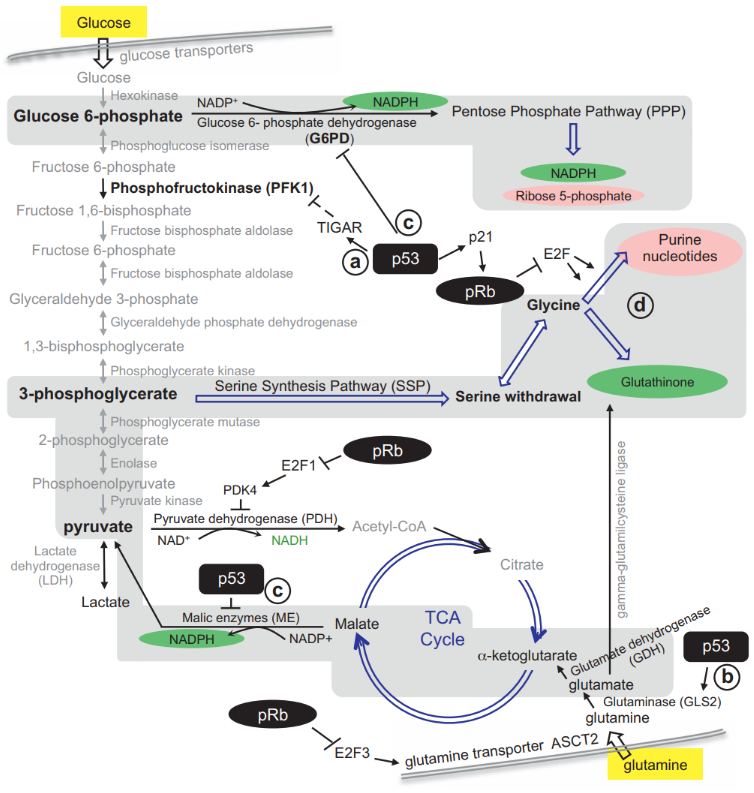
Tanto p53 como pRb pueden tener efectos conjuntos apreciables. Un ejemplo es su acción en el metabolismo del cáncer, ya que regulan el anabolismo de múltiples moléculas o en la defensa antioxidante (para más información sobre la importancia del metabolismo en el cáncer pincha aquí).
Para saber más:
- Tumor Suppressor Genes. Science 5035, 254, 1138-1146 (1991).
- MicroRNA-34 family: a potential tumor suppressor and therapeutic candidate in cancer. J Exp Clin Cancer Res 38, 53 (2019).
Bibliografía empleada para elaborar el texto y las imágenes:
Flores M, Goodrich DW. Retinoblastoma protein paralogs and tumor suppression. Frontiers in Genetics 13 (2022).
Hanahan D, Weinberg RA. The hallmarks of cancer. Cell 100, 57-70 (2000).
Hanahan D, Weinberg RA. Hallmarks of cancer: the next generation. Cell 144, 646-674 (2011).
Mantovani F, Collavin L, Del Sal G. Mutant p53 as a guardian of the cancer cell. Cell Death Differ 26, 199–212 (2019).
Negrini S, Gorgoulis V, Halazonetis T. Genomic instability — an evolving hallmark of cancer. Nat Rev Mol Cell Biol 11, 220–228 (2010).
Wang H, Guo M, Wei H et al. Targeting p53 pathways: mechanisms, structures, and advances in therapy. Sig Transduct Target Ther 8, 92 (2023).
Zhu L, Lu Z, Zhao H. Antitumor mechanisms when pRb and p53 are genetically inactivated. Oncogene 34, 4547–4557 (2015).